艾尼瓦尔·塔力甫熊成热甫哈提·赛买提玉苏甫·买提努尔吐尔逊·吾甫尔阿肯木江·艾尔肯居来提·阿不都瓦衣提买买提艾力·卡的摘要
目的
通过生物信息学方法探索与白癜风进展相关的信号通路和基因。
方法
从GEO数据库中下载白癜风芯片检测数据集GSE75819,利用R语言软件中limma包的LMFit和eBayes函数筛选15例印度白癜风患者皮损与非皮损组织间的差异表达基因(DEG)。通过京都基因与基因组数据库(KEGG)、基因本体论(GO)分析和基因集富集分析(GSEA)评估DEG的富集途径和功能。通过蛋白-蛋白相互作用网络从DEG中筛选中心基因。2019年1 - 6月于新疆维吾尔自治区维吾尔医医院收集8例汉族寻常型白癜风患者皮损及非皮损皮肤组织标本,采用实时定量PCR法验证上述上调及下调差异最大的10个DEG的表达。
结果
与15例非皮损组织相比,在15例白癜风皮损组织中共发现148个DEG,其中KRT9、CXCL10、C8ORF59、TPSAB1、RPL26为前5位上调基因,SILV、RPPH1、TYRP1、MLANA、LOC401115为前5位下调基因,且经实时定量PCR在8例汉族白癜风患者皮损及非皮损组织中验证。GO分析显示,DEG主要富集于翻译起始、细胞对脂多糖的反应、核糖体、核糖体亚基和核糖体的结构组成等。KEGG通路分析显示,DEG主要富集于酪氨酸代谢、PPAR信号通路、氧化磷酸化和Toll样受体信号通路。蛋白-蛋白相互作用分析筛选出UPF3B、SNRPG、MRPL13和RPL26L1 4个中心基因。
结论
KRT9、CXCL10、C8ORF59、TPSAB1、RPL26、SILV、RPPH1、TYRP1、MLANA及LOC401115可能作为白癜风潜在的诊断标记分子和治疗靶点。
白癜风是一种自身免疫性疾病,也是一种多基因遗传性疾病,由遗传易感性个体的免疫细胞与环境的相互作用引起[[url=]1[/url]]。本研究通过生物信息学方法分析GEO(gene expression omnibus)数据集,寻找与白癜风进展相关的基因,并在中国汉族白癜风患者中验证差异表达基因(differentially expressed genes,DEG)。
一、对象与方法
1.基因数据集分析:
从GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)中选择编号为GSE75819数据集。该数据来源于15例印度寻常型白癜风患者,包含白癜风皮损组织15例(白癜风皮损组)及非皮损皮肤组织15例(正常对照组)[[url=]2[/url]]。其中,男4例、女11例;1例年龄未提供,另14例年龄10 ~ 45(22.50 ± 2.20)岁;8例白癜风皮损组织来自小腿,3例来自前臂,来自膝盖、手腕、前额、颈部的皮损组织各1例,而15例非皮损组织均来自臀部。
(1)DEG的鉴定:首先对全部基因进行主成分分析,以压缩数据空间,将多元数据的特征在低维空间里直观地表示出来。利用R语言软件limma包(version 3.32.3)中LMFit和eBayes函数筛选上述白癜风皮损组织与非皮损组织间的DEG。当P < 0.05且|log2(差异倍数)| > 1.5时认为该基因为DEG。使用R软件ggplot2包(http://cran.r-project.org/web/packages/gplots/index.html)构建DEG的火山图和聚类分析图。
(2)DEG富集分析:通过基因本体论(gene ontology,GO)及京都基因和基因组(Kyoto encyclopedia of genes and genomes,KEGG)数据库分析DEG的功能。
GO功能富集分析:利用R语言软件clusterProfiler对DEG进行GO功能富集分析,包含生物过程、细胞成分和分子功能3个方面。P < 0.05认为显著富集。
KEGG通路富集分析:应用Cytoscape软件插件BinGO对DEG进行KEGG通路分析。P < 0.05认为是显著富集的通路。
基因集富集分析(gene set enrichment analysis,GSEA):对DEG进行GSEA(http://www.broadinstitute.org/gsea)、基因集GO功能分析以及KEGG通路分析,以分析DEG显著涉及的基因功能和代谢途径。错误发现率(false discovery rate,FDR)< 25%的基因集被认为是显著富集的基因集。
(3)蛋白-蛋白相互作用分析:应用STRING数据库(https://string-db.org/)对白癜风皮损组织与非皮损组织间的DEG进行蛋白-蛋白相互作用分析,交互评分最低要求为0.7,鉴定与白癜风相关的中心基因。应用Cytoscape软件可视化蛋白-蛋白相互作用网络。
2.皮肤组织样本收集:
2019年1 - 6月于新疆维吾尔自治区维吾尔医医院收集8例汉族寻常型白癜风患者皮损及非皮损皮肤组织标本,男1例,女7例,年龄10 ~ 25(15.25 ± 5.12)岁,发病年龄9 ~ 18(13.13 ± 2.85)岁。4例为进展期(皮损正在进展或6个月内出现新皮损),4例为稳定期(6个月内皮损大小/数量未增加)。4例皮损组织取自小腿,3例取自前臂,1例来自颈部;8例非皮损组织均取自臀部。所有患者在纳入本研究前3个月以上未接受任何全身或局部治疗。本研究经新疆维吾尔自治区维吾尔医医院伦理委员会批准(伦理审批编号:LCEC[2016]001),所有患者均签署了知情同意书。
3.实时定量PCR验证DEG mRNA的表达:
使用RNAiso-Plus试剂盒[宝生物工程(大连)有限公司]提取8例白癜风患者皮损及非皮损组织中的总RNA。采用cDNA合成试剂盒[宝生物工程(大连)有限公司]将RNA逆转录为cDNA。以1 μg cDNA为底物,应用SYBR Green试剂(美国ThermoFisher Scientific公司),在ABI 7300 Real-Time PCR系统(美国Applied Biosystems公司)上行实时定量PCR检测,引物序列见[url=]表1[/url]。以甘油醛-3-磷酸脱氢酶(GAPDH)为内参,用2-∆∆Ct方法计算DEG mRNA的相对表达量,其中∆Ct=目的基因Ct值-内参Ct值,∆∆Ct=白癜风组∆Ct值-对照组∆Ct值。


表1
实时定量PCR中引物序列
4.统计分析方法:
应用SPSS21.0软件包进行统计分析。采用两独立样本t检验比较白癜风皮损组织与非皮损组织中DEG mRNA的表达。P < 0.05认为差异有统计学意义。
二、结果
1.白癜风皮损与非皮损皮肤组织间DEG的分析与鉴定:
主成分分析显示,15例白癜风皮损与非皮损组织基本分离,区分度较好,见[url=]图1A[/url]。R语言软件limma包筛选DEG显示,与非皮损对照组相比,白癜风皮损组共发现148个DEG,其中22个基因表达下调,126个基因表达上调([url=]图1B[/url])。[url=]图1C[/url]为148个DEG的聚类分析图。其中,角蛋白9(keratin 9,KRT9)、C-X-C趋化因子配体10(C-X-C motif chemokine ligand 10,CXCL10)、核糖体生物起源因子(ribosomal biogenesis factor,C8ORF59)、类胰蛋白酶α/β-1(tryptase alpha/beta-1,TPSAB1)和核糖体蛋白L26(ribosomal protein L26,RPL26)是前5位上调的基因;前黑素体蛋白(premelanosome protein,SILV)、核糖核酸酶P RNA组分H1(ribonuclease P RNA component H1,RPPH1)、酪氨酸酶相关蛋白1(tyrosinase related protein 1,TYRP1)、黑色素A(melan-A,MLANA)和4号染色体开放阅读框48(chromosome 4 open reading frame 48,LOC401115)是前5个下调基因。

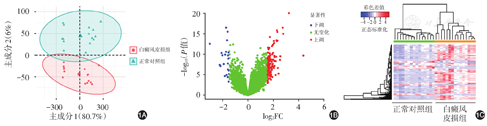
图1
15例白癜风患者皮损与非皮损皮肤组织间差异表达基因(DEG)分析 1A:主成分分析图,两组区分度较好;1B:白癜风皮损与非皮损组织间DEG的火山图;1C:DEG的聚类分析热图
实时定量PCR验证8例白癜风皮损与非皮损皮肤组织间10个上调及下调基因显示,与非皮损组织相比,皮损中KRT9、CXCL10、C8ORF59、TPSAB1和RPL26的mRNA表达显著升高(t值分别为11.99、11.92、11.59、9.91、10.58,均P < 0.001),而SILV、RPPH1、TYRP1、MLANA和LOC401115的mRNA表达显著降低(t值分别为12.00、10.38、12.70、12.98、10.14,均P < 0.001),见[url=]图2[/url],进一步证实了上述生物信息学分析结果。

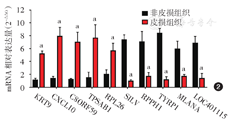
图2
实时定量PCR检测8例白癜风患者皮损与非皮损组织间差异表达基因KRT9、CXCL10、C8ORF59、TPSAB1、RPL26、SILV、RPPH1、TYRP1、MLANA和LOC401115的mRNA相对表达 与非皮损组织相比,白癜风皮损组织中KRT9、CXCL10、C8ORF59、TPSAB1和RPL26的mRNA表达显著升高,而SILV、RPPH1、TYRP1、MLANA和LOC401115的mRNA表达显著降低,a:P < 0.001
2.DEG的富集分析:
GO分析显示,在生物过程方面,148个DEG主要富集于翻译起始、细胞对脂多糖的反应、发育性色素沉着和细胞对细菌起源分子的反应。细胞成分方面,148个DEG主要富集在核糖体、核糖体亚基和线粒体内膜中。分子功能方面,148个DEG富集在核糖体的结构成分中。
KEGG途径分析显示,DEG在酪氨酸代谢、Toll样受体(Toll-like receptor,TLR)信号通路、肿瘤坏死因子(tumor necrosis factor,TNF)信号通路、过氧化物酶体增殖物激活受体(peroxisome proliferator activated receptor,PPAR)信号通路、氧化磷酸化等方面均显著富集。
GSEA GO分析显示,与非皮损组织相比,白癜风皮损组织中462个基因集显著富集(FDR值< 25%),包括460个上升,2个下降。其中突触信号、细胞膜蛋白复合物、磷脂酶C激活G蛋白偶联受体信号通路上调幅度最大,神经系统过程负调控、CXCR趋化因子受体结合、淋巴细胞趋化调节下调幅度最大。GSEA KEGG分析显示,31个基因集显著富集(FDR值< 25%),均上调,包括细胞黏附分子Cams、钙信号通路、黑素瘤、白细胞跨内皮迁移、JAK/STAT信号通路和自然杀伤细胞介导的细胞毒性。
3.DEG的蛋白-蛋白相互作用分析:
DEG中mRNA衰变调节因子UPF3B(UPF3B regulator of nonsense mediated mRNA decay,UPF3B)、小核核糖核蛋白多肽G(small nuclear ribonucleoprotein polypeptide G,SNRPG)、线粒体核糖体蛋白L13(mitochondrial ribosomal protein L13,MRPL13)和核糖体蛋白L26L1(ribosomal protein L26 like 1,RPL26L1)基因在蛋白-蛋白相互作用网络中得分最高。
三、讨论
白癜风的发病机制复杂,目前已发现遗传易感性、代谢异常、环境变化以及炎症和免疫反应的改变与其发病有关[[url=]3[/url],[url=]4[/url],[url=]5[/url]]。Singh等[[url=]2[/url]]通过高通量测序技术对比分析15例白癜风患者皮损和非皮损皮肤组织的全基因组,发现1 705个DEG,包括786个上调基因和919个下调基因,进一步分析发现,这些DEG主要富集于黑色素生成途径、角化及角质细胞分化途径。本研究中,我们首次利用R语言limma包对该数据集进行了更为深入的探究,同时借助实时定量PCR实验在另8例中国汉族白癜风患者中验证,试图更为深入地揭示白癜风的发病机制。微阵列数据分析显示,与15例白癜风非皮损组织相比,皮损组织中发现148个DEG,包含126个上调基因和22个下调基因。
本研究显示,KRT9、CXCL10、C8ORF59、TPSAB1和RPL26是白癜风皮损组织中前5位上调的基因,而SILV、RPPH1、TYRP1、MLANA和LOC401115是前5位下调的基因,随后通过实时定量PCR在8例白癜风患者皮损和非皮损组织中得到了验证。KRT9位于染色体17q21上,在维持掌跖表皮的完整性方面起重要作用[[url=]6[/url]]。研究显示,KRT9基因突变与表皮松解性掌跖角化病的发病密切相关[[url=]7[/url]],但其在白癜风发展中的作用仍不清楚。邓莉等[[url=]8[/url]]研究显示,进展期白癜风患者血清CXCL10水平明显高于健康对照者,而本研究结果亦显示,CXCL10基因在白癜风皮损组织中表达上调。SILV基因被认为在黑素小体的发育过程中起重要作用,其在黑素瘤患者中过表达,被认为是黑素瘤的诊断标志物和治疗靶点,而SILV的表达下调可能导致白癜风患者皮肤部分脱色[[url=]9[/url]]。此外,Cui等[[url=]10[/url]]研究表明,酪氨酸酶和TYRP1复合物(Tyr1-TYRP1)在黑素中间体的合成中发挥重要作用,可调节活性氧的产生,促进白癜风的发生。在墨西哥寻常型白癜风患者中TYRP1和MLANA表达升高[[url=]11[/url]],但这与我们的研究结果相反,尚需进一步研究。
GO分析显示,这些DEG主要富集于翻译起始、细胞对脂多糖的反应、发育性色素沉着、细胞对细菌起源分子的反应、核糖体、核糖体亚基、线粒体内膜以及核糖体的结构成分中。KEGG分析显示,这些DEG主要富集于酪氨酸代谢、PPAR信号通路、氧化磷酸化、TLR和TNF信号通路等,提示酪氨酸代谢、TLR、TNF信号通路等在白癜风进展中的潜在作用。唐亚平等[[url=]12[/url]]研究显示,白癜风患者TLR7、髓样分化因子88(myeloid differentiation factor88,MyD88)及NF-κB mRNA的表达高于健康对照组,而TLR9 mRNA的表达低于健康对照组,表明TLR7、TLR9及其调控通路中的关键信号分子NF-κB可能参与白癜风的发病。
利用蛋白-蛋白相互作用网络分析,我们从148个DEG中识别出4个中心基因,包括UPF3B、SNRPG、MRPL13和RPL26L1。UPF3B位于X染色体上,其编码蛋白作用于无义介导的mRNA衰减通路,该通路在调节基因表达方面有重要意义[[url=]13[/url]]。目前研究表明,UPF3B突变与染色体X相关性智力障碍和神经发育障碍有关[[url=]14[/url]]。SNRPN是印迹基因簇的成员之一,通常由父系遗传的染色体表达,在神经发育中发挥重要作用[[url=]15[/url]]。MRPL13是一种线粒体核糖体蛋白,其基因缺失可导致线粒体DNA的缺失和线粒体编码蛋白能力的丧失。Wang等[[url=]16[/url]]发现,MRPL13的高表达与乳腺癌患者的不良预后有关。RPL26L1属于核糖体蛋白家族,与核糖体蛋白L26具有高度的序列相似性,并在蛋白质合成中发挥作用。研究显示,RPL26L1在系统性红斑狼疮患者中表达升高,可能参与了系统性红斑狼疮的发病[[url=]17[/url]]。由于白癜风与系统性红斑狼疮均与炎症反应相关[[url=]18[/url]],推测RPL26L1可能在白癜风进展中发挥作用,但还需通过实验进一步验证。
综上,本研究显示KRT9、CXCL10、TPSAB1、SILV、TYRP1、MLANA、UPF3B、SNRPG、MRPL13、RPL26L1等基因在白癜风皮损组织中差异表达,揭示其在白癜风诊断和治疗中的潜在应用价值,但其在白癜风发病中的具体作用机制还有待通过体内体外实验进一步验证。
利益冲突
所有作者均声明不存在利益冲突
参考文献
[1]
章一近,许爱娥.白癜风相关基因研究进展[J].国外医学(皮肤性病学分册), 2004,30(2):91-94. doi: 10.3760/cma.j.issn.1673-4173.2004.02.008.
[2]
SinghA, GotherwalV, JunniP, et al. Mapping architectural and transcriptional alterations in non-lesional and lesional epidermis in vitiligo[J]. Sci Rep, 2017,7(1):9860. doi: 10.1038/s41598-017-10253-w.
[3]
杨丽,陆东庆.白癜风发病机制中相关细胞因子的研究进展[J].皮肤性病诊疗学杂志, 2016,23(1):68-70,76. doi: 10.3969/j.issn.1674-8468.2016.01.021.
[4]
卢保振,柏冰雪.调节性T细胞在白癜风发病中的研究进展[J].国际免疫学杂志, 2016,39(1):93-96. doi: 10.3760/cma.j.issn.1673-4394.2016.01.021.
[5]
许爱娥.白癜风发病机制及治疗靶点的研究现状[J].中华皮肤科杂志, 2016,49(6):377-379. doi: 10.3760/cma.j.issn.0412-4030.2016.06.001.
[6]
毛薇,何新辉,张咸宁,等.三个表皮松解性掌跖角化症家系KRT9基因突变的研究[J].中华皮肤科杂志, 2004,37(9):503-505. doi: 10.3760/j.issn:0412-4030.2004.09.001.
[7]
LiuX, QiuC, HeR, et al. Keratin 9 L164P mutation in a Chinese pedigree with epidermolytic palmoplantar keratoderma, cytokeratin analysis, and literature review[J]. Mol Genet Genomic Med, 2019,7(11):e977. doi: 10.1002/mgg3.977.
[8]
邓莉,江雪,王禹毅,等. CXCL10、CXCR3及T细胞亚群在进展期白癜风患者外周血中的表达及复方中药对其影响[J].重庆医学, 2018,47(2):164-166. doi: 10.3969/j.issn.1671-8348.2018.02.006.
[9]
KuehnC, WeikardR. Multiple splice variants within the bovine silver homologue(SILV)gene affecting coat color in cattle indicate a function additional to fibril formation in melanophores[J]. BMC Genomics, 2007,8:335. doi: 10.1186/1471-2164-8-335.
[10]
CuiTT, YiXL, ZhangWG, et al. MiR-196a-2 rs11614913 polymorphism is associated with vitiligo by affecting heterodimeric molecular complexes of Tyr and Tyrp1[J]. Arch Dermatol Res, 2015,307(8):683-692. doi: 10.1007/s00403-015-1563-1.
[11]
Salinas-SantanderM, TrevinoV, De la Rosa-MorenoE, et al. CAPN3, DCT, MLANA and TYRP1 are overexpressed in skin of vitiligo vulgaris Mexican patients[J]. ExpTher Med, 2018,15(3):2804-2811. doi: 10.3892/etm.2018.5764.
[12]
唐亚平,盛文婷,周欣,等. Toll样受体7、9及其调控分子MyD88、NF-κB在白癜风患者外周血单个核细胞中的表达[J].中华皮肤科杂志, 2017,50(1):13-17. doi: 10.3760/cma.j.issn.0412-4030.2017.01.004.
[13]
KervestinS, JacobsonA. NMD: a multifaceted response to premature translational termination[J]. Nat Rev Mol Cell Biol, 2012,13(11):700-712. doi: 10.1038/nrm3454.
[14]
AlrahbeniT, SartorF, AndersonJ, et al. Full UPF3B function is critical for neuronal differentiation of neural stem cells[J]. Mol Brain, 2015,8:33. doi: 10.1186/s13041-015-0122-1.
[15]
LiH, ZhaoP, XuQ, et al. The autism-related gene SNRPN regulates cortical and spine development via controlling nuclear receptor Nr4a1[J]. Sci Rep, 2016,6:29878. doi: 10.1038/srep29878.
[16]
WangK, LiL, FuL, et al. Integrated bioinformatics analysis the function of RNA binding proteins(RBPs)and their prognostic value in breast cancer[J]. Front Pharmacol, 2019,10:140. doi: 10.3389/fphar.2019.00140.
[17]
YangF, ZhaiZ, LuoX, et al. Bioinformatics identification of key candidate genes and pathways associated with systemic lupus erythematosus[J]. ClinRheumatol, 2020,39(2):425-434. doi: 10.1007/s10067-019-04751-7.
[18]
RashighiM, HarrisJE. Vitiligo pathogenesis and emerging treatments[J]. DermatolClin, 2017,35(2):257-265. doi: 10.1016/j.det.2016.11.014.